במחקר שפורסם לאחרונה בכתב העת טֶבַעצוות גדול של חוקרים מארצות הברית (ארה"ב) השתמש ברצף חומצה ריבונוקלאית (RNA) חד-תאית בשילוב עם הכלאה של פלואורסצנטי ברזולוציה גבוהה באתרו כדי לקבוע את זהותם של סוגי התאים השונים המתואמים במרחב כדי להוליד את המתחם מבנה מורפולוגי של הלב.
מחקר: קהילות סלולריות מאורגנות במרחב יוצרות את הלב האנושי המתפתח. קרדיט תמונה: beerkoff / Shutterstock
רקע כללי
לכל מבנה מורכב של הלב האנושי יש תפקידים ספציפיים התורמים לתפקוד לבבי יעיל, וההפסקה של כל אחד מהתפקודים הללו עלולה להוביל לליקויים מולדים כגון מחלת לב מולדת בילדים ומחלות לב כגון valvulopathies וקרדיומיופתיה אצל מבוגרים. עם זאת, למרות התפקיד הקריטי של הלב בגוף האדם, הארגון והתפקוד של מבני הלב והאופן שבו הם מתקשרים זה עם זה עדיין לא מובנים.
לגבי המחקר
במחקר הנוכחי, החוקרים השתמשו בגישת ריצוף RNA חד-תא (scRNAseq) יחד עם פלואורסצנציה חזקה מרובת שגיאות באתר הכלאה (MER-FISH). אסטרטגיה זו אפשרה להם לשלב תעתיקי תא בודד וביולוגיה מרחבית ולהמחיש, לנתח ולכמת את תעתיקי ה-RNA של מספר רב של גנים מתא בודד.
הם התחילו בזיהוי שושלות התאים שהיו חלק מהלב המתפתח, מה שעזר לקבוע כיצד סוגי תאי הלב השונים מתאספים למבנים מורכבים ומתואמים כדי לווסת את תפקוד הלב האנושי. ה-scRNAseq נערך בשכפולים ונותח עבור לבבות אנושיים בשלבי צמיחה שונים, החל מתשעה שבועות ועד 16 שבועות לאחר ההתעברות.
התאים הבודדים שהתקבלו, מעל 140 מיליון במספר, סווגו באופן תעתיק לחמישה תאים: קרדיומיוציטים, אנדותל, מזנכימלי, עצבי ודם. בתוך תאים אלה, ניתוח של סמני גנים עזר לזהות 12 מחלקות תאים, עם ניתוחי מקבץ שלאחר מכן לזהות 39 אוכלוסיות ו-75 תת-אוכלוסיות של תאים.
לאחר מכן נעשה שימוש ב-MER-FISH למיפוי מרחבי של תאי הלב ולחקור את המנגנונים התאיים שדרכם כוונו העיצוב מחדש והמורפוגנזה של הלב, כולל התפתחות דופן החדר. ארגון התאים שזוהו באמצעות scRNAseq, במיוחד בתקופות התפתחות כמו דחיסה של דופן שריר הלב, נחקר באמצעות הדמיית MER-FISH.
לאחר מכן מטרת המחקר הייתה לפענח את ההרכבה של תאים קרדיו-וסקולריים ספציפיים אלו לשכונות הסלולריות המתחברות יחד ליצירת מבנים רב-תאים התורמים לתפקוד הלב. המדענים גם חקרו את המורכבות הארגונית והתאית של אזורים ספציפיים, כגון החדרים, על ידי חקר התאים בתוך החדרים שזוהו, בודדו ומפו באמצעות MER-FISH. בנוסף, נעשה שימוש במודלים של עכברים כדי לחקור את האינטראקציות בין תאים באמצעות in vivo ניסויים, ותאי גזע פלוריפוטנטיים מבני אדם שימשו להערכת אותם ב בַּמַבחֵנָה ניסויים.
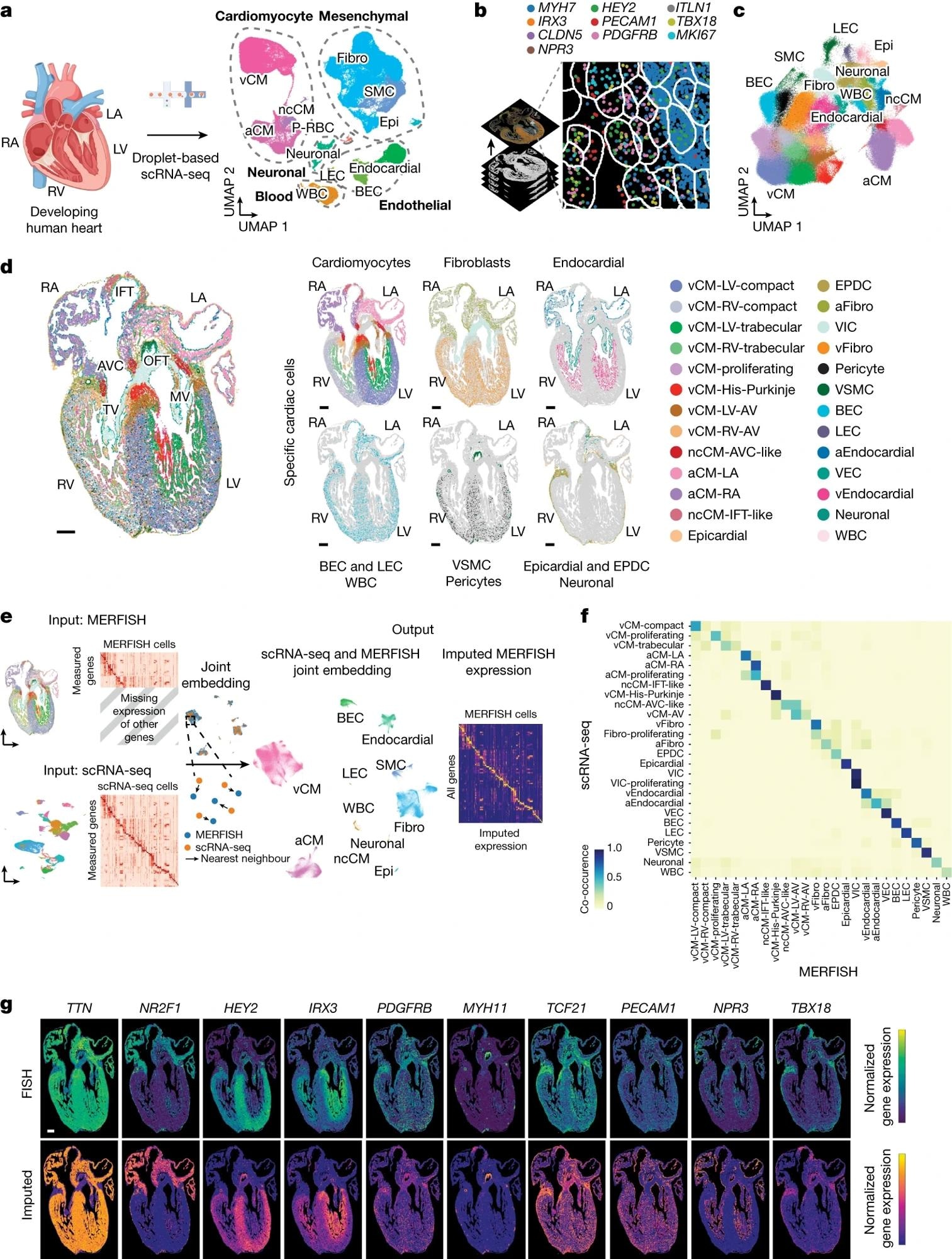 א, משמאל, סכמטי של הניסוי. מימין, scRNA-seq מזהה מגוון מגוון של תאי לב נפרדים היוצרים את הלב האנושי המתפתח כפי שמוצג על ידי קירוב והקרנה סעפת אחידה (UMAP) של ~143,000 תאים. ב, סכמטי מראה כיצד 238 גנים ספציפיים לתאים לבביים זוהו באופן מרחבי באמצעות MERFISH. נקודות בצבע פסאודו מסמנות את מיקומן של מולקולות בודדות של עשרה תעתיקי RNA ספציפיים. גכ-250,000 תאי לב המזוהים עם MERFISH התקבצו לאוכלוסיות תאים ספציפיות כפי שמוצג על ידי UMAP ונצבעו בהתאם ב ד. דתאי MERFISH מזוהים מופו באופן מרחבי על פני קטע קדמי של לב של 13 pcw (משמאל) והוצגו לפי קבוצות תאים עיקריות (מימין). ההטמעה משותפת בין MERFISH ומערכות scRNA-seq תואמות גיל אפשרה העברת תווית תאים וזקיפת גנים של MERFISH. ומפת חום של Co-Currence מציגה את ההתאמה של הערות התא של תאי MERFISH לאלו שהועברו ממערך הנתונים של 13 pcw scRNA-seq. ז, ביצועי זקיפת גנים אומתו באופן מרחבי על ידי השוואת פרופילי ביטוי גנים מנורמלים של גנים סמנים שנמדדו על ידי MERFISH עם פרופילי ביטוי גנים זקופים המקבילים. אפי, אפיקרד; MV, שסתום מיטרלי; P–RBC, טסיות דם–כדוריות דם אדומות; טלוויזיה, שסתום תלת-צדדי. סרגל קנה מידה, 250 מיקרומטר (ז). איור ב א נוצר באמצעות BioRender (https://www.biorender.com).
א, משמאל, סכמטי של הניסוי. מימין, scRNA-seq מזהה מגוון מגוון של תאי לב נפרדים היוצרים את הלב האנושי המתפתח כפי שמוצג על ידי קירוב והקרנה סעפת אחידה (UMAP) של ~143,000 תאים. ב, סכמטי מראה כיצד 238 גנים ספציפיים לתאים לבביים זוהו באופן מרחבי באמצעות MERFISH. נקודות בצבע פסאודו מסמנות את מיקומן של מולקולות בודדות של עשרה תעתיקי RNA ספציפיים. גכ-250,000 תאי לב המזוהים עם MERFISH התקבצו לאוכלוסיות תאים ספציפיות כפי שמוצג על ידי UMAP ונצבעו בהתאם ב ד. דתאי MERFISH מזוהים מופו באופן מרחבי על פני קטע קדמי של לב של 13 pcw (משמאל) והוצגו לפי קבוצות תאים עיקריות (מימין). ההטמעה משותפת בין MERFISH ומערכות scRNA-seq תואמות גיל אפשרה העברת תווית תאים וזקיפת גנים של MERFISH. ומפת חום של Co-Currence מציגה את ההתאמה של הערות התא של תאי MERFISH לאלו שהועברו ממערך הנתונים של 13 pcw scRNA-seq. ז, ביצועי זקיפת גנים אומתו באופן מרחבי על ידי השוואת פרופילי ביטוי גנים מנורמלים של גנים סמנים שנמדדו על ידי MERFISH עם פרופילי ביטוי גנים זקופים המקבילים. אפי, אפיקרד; MV, שסתום מיטרלי; P–RBC, טסיות דם–כדוריות דם אדומות; טלוויזיה, שסתום תלת-צדדי. סרגל קנה מידה, 250 מיקרומטר (ז). איור ב א נוצר באמצעות BioRender (https://www.biorender.com).
תוצאות
הממצאים גילו שסוגי תאי לב שונים השתייכו לתת-אוכלוסיות ספציפיות שהיו חלק מקהילות ספציפיות, כאשר ההתמחות התפקודית מוגדרת בהתאם לאזור האנטומי שבו הם נמצאים ולמערכת האקולוגית התאית. שושלות הקרדיומיוציטים היו תא התאים הגדול ביותר שזוהה באמצעות MER-FISH. המחקר מצא גם שתאים שהשתייכו לתאים שאינם קרדיומיוציטים עברו הפרדה לאוכלוסיות ותת-אוכלוסיות ותרמו להיווצרות מבנים ואזורים ספציפיים בלב.
תת-אוכלוסיות קרדיומיוציטים באזורי החדרים הראו יכולת לבנות מבנים למינליים מורכבים בדופן החדר וליצור קהילות תאיות עם תת-אוכלוסיות אחרות של תאי לב. יתר על כן, ה in vivo ו בַּמַבחֵנָה ניסויים שנערכו כדי להבין את האינטראקציות בין תאים גילו שהארגון המרחבי של תת-אוכלוסיות של תאי לב במהלך המורפוגנזה של דופן החדר נערך דרך מסלולי איתות שונים.
המחקר מצא גם אזורי לב המורכבים משילובים מאורגנים מרחבית של אוכלוסיות תאים שנפרדו יחד, הנקראות קהילות סלולריות. קהילות תאיות אלו היו שונות במספר ובסוגי אוכלוסיות התאים, ובתוך קהילות אלו הוגדרו שכנים של כל תא לב ברדיוס של 150 מיקרומטר. לאוכלוסיות התאים המקיימות אינטראקציה אלו היו גם מסלולי איתות סלולריים ברורים.
מסקנות
בסך הכל, המחקר מצא שקרדיומיוציטים היו המדור הגדול ביותר של סוגי תאים בלב המתפתח, וכל סוגי התאים הציגו התפלגות מבנית ואזורית ברורה בלב. אוכלוסיות תאים ספציפיות יצרו גם קהילות תאיות בשילובים שונים, כאשר מסלולי איתות בין אוכלוסיות התאים בתוך הקהילה הגדירו את המבנה והתפקוד שלהן. המחקר סייע בהבנת התפתחות המבנה המורכב של הלב האנושי, והעניק דרכים פוטנציאליות לטיפול במחלות לב מבניות.